 Hvorfor Darwinismen ikke kan fullende innovasjon eller forklare opprinnelser
Hvorfor Darwinismen ikke kan fullende innovasjon eller forklare opprinnelser
Ann Gauger; 13. februar 2018
Oversatt herfra.
Bilde 1: E. coli bakterier, ved NIAID [ CC BY 2.0 ], via Wikimedia Commons.
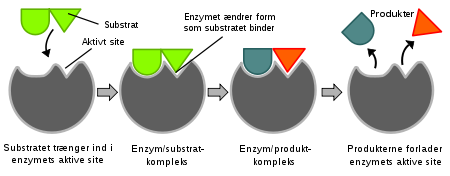
Når man ønsker å modifisere et enzym for en ny funksjon, som Matti Leisola forklarer i kapittel 10 i sin nye bok Heretic, er det to måter å gå fram på.
For det første kan man bruke det som kalles styrt evolusjon. Begrepet er litt misvisende - det ligner på evolusjon, fordi det innebærer å generere millioner av tilfeldige mutasjoner i genet som studeres (ikke et enkelt gen, men millioner av kopier av det genet), og deretter velge for ønsket funksjon ved å sette de muterte gener i bakterier og be de resulterende proteinene om å utføre den nye funksjonen.
 Men det er ikke i det hele tatt som ekte darwinistisk evolusjon, ved at eksperimentøren bestemmer hva det overordnede målet er (evolusjonen har ingen mål) og lager en plan for hvordan man skal oppnå det. Leder av eksperimentet bestemmer mutasjonshastigheten og forårsaker at alle disse mutasjonene finner sted i en populasjon av bakterier, samtidig, og screeningen er også designet av eksperimentet for å sortere gjennom alle de millioner av mutasjoner for ønsket , planlagt endring.
Men det er ikke i det hele tatt som ekte darwinistisk evolusjon, ved at eksperimentøren bestemmer hva det overordnede målet er (evolusjonen har ingen mål) og lager en plan for hvordan man skal oppnå det. Leder av eksperimentet bestemmer mutasjonshastigheten og forårsaker at alle disse mutasjonene finner sted i en populasjon av bakterier, samtidig, og screeningen er også designet av eksperimentet for å sortere gjennom alle de millioner av mutasjoner for ønsket , planlagt endring.
For det andre kan man bruke rasjonelt design som, som navnet antyder, er et forsøk på å skifte proteinets funksjon ved å studere hvordan det virker, og gjøre endringer i proteinets sekvens. Utforskeren håper disse endringene vil gjøre den ønskede funksjonelle endringen. å forstå hvilke forandringer som skal fungere, krever mye kunnskap om det aktuelle enzymets kjemi, så vel som dets tredimensjonale struktur. Men selv med all den kunnskapen er det ingen garanti for at en bestemt forandring vil fungere som planlagt. Mye prøving og feiling er involvert, fordi enzymstruktur og funksjon er temmelig kompliserte.
Jeg har brukt begge disse teknikkene i laboratoriet, men jeg er ikke i Mattis klasse. Han er en mester på å endre enzymer. Og én ting vet han: å få ny enzymatisk funksjon vil ikke skje ved tilfeldig mutasjon og naturlig utvalg. Han har tilbrakt tiår med å jobbe i laboratoriet for å konstruere enzymer og kjenner utfordringene ganske bra. I kapittel 10 legger han fram bevisene for hvorfor tilfeldig variasjon og naturlig utvalg, eller gen duplisering og nøytral evolusjon for å si det i andre termer, ikke vil fungere.
For det første er foldede funksjonelle proteiner ekstremt sjeldne i sekvensrommet. Dette har blitt vist av flere laboratorier ved å måle mutasjons-følsomheten til forskjellige enzymer og bruke det til å estimere hvilken brøkdel av alle mulige sekvenser som kan utføre hvert enzyms funksjon. For det andre er det bevis som viser at å få et helt nytt protein fra en eksisterende -her"er usannsynlig at det  skjer ved evolusjon gjennom en sti med foldede mellomliggende sekvenser." Videre, til og med et enzym med lignende struktur, men ingen delt kjemi med et annet enzym, kan ikke bli konvertert til å utføre funksjonen til det tilsynelatende relaterte enzymet.
skjer ved evolusjon gjennom en sti med foldede mellomliggende sekvenser." Videre, til og med et enzym med lignende struktur, men ingen delt kjemi med et annet enzym, kan ikke bli konvertert til å utføre funksjonen til det tilsynelatende relaterte enzymet.
En del av problemet er at det kombinatoriske problemet er så stort. Matti beskriver det å delta på et PhD-forsvar for en student som ikke hadde kunnet fullføre et bestemt protein-re-engineering problem ved hjelp av enten rettet evolusjon eller rasjonelt design. Matti spurte ham om han hadde beregnet oddsene for å finne den rette kombinasjonen av mutasjoner når bare to eller tre spesifikke endringer var nødvendig. Siden studenten ikke hadde det, foreslo Matti at oddsen oversteg tjue millioner til en. En annen måte, Marci Reeves, Doug Axe og jeg har beregnet at å få fire mutasjoner ville ta (10^15) år for E. coli -her, ved hjelp av sjenerøse estimater for populasjonsstørrelse, mutasjonsrate, doblingstid, etc.
Det er sant at i laboratoriet kan proteiningeniører forbedre termisk stabilitet, endre pH-stabilitet, forbedre sidereaksjoner og øke enzymaktiviteten ved tilfeldige turer. Men alt dette innebærer en stor del av utøver-engasjement. Og det er begrensninger. Det kan ikke skje hvor tre eller flere mutasjoner må skje på en gang. Det må være en valgbar sti hele veien. Og man må kunne lage nok mutanter og skjerme dem raskt for å ha et håp om å finne de svært sjeldne som fungerer.
Det er mulig å konstruere en ny vei inn i organismer, det vil si hvis man velger en organisme som kan tolerere endrede innføringer. Metabolismen som helhet kan kastes ut av balanse dersom en ny aktivitet blir introdusert uten riktig omsorg. Matti beskriver hvordan hans laboratorium med suksess har opprettet en gjærstamme som var i stand til å produsere xylitol, som de fikk patent på. Men det krever at to nye enzymer blir introdusert, og mye arbeid og planlegging fra forskernes side. Evolusjonen har ikke evnen til å planlegge. Den har intet fremsyn.
Det er også sant at noen ganger vil det oppstå en spontan mutasjon som vil gi en ny evne til en organisme. Et eksempel var når Aerobacter aerogenes utviklet evnen til å bruke xylitol som en karbonkilde. Det viste seg at dette ikke var utviklingen av en ny enzymevne, men snarere brudd på et regulatorisk element -her, noe som resulterte i den essensielle påvirkningen av et enzym som allerede eksisterer. Tilsvarende utviklet E. coli i Lenskis laboratorium muligheten til å bruke sitrat som en karbonkilde, som følge av DNA-omlegging. Det viste seg også å være å slå på et gen som normalt var avstengt.
 Endelig viste utviklingen av enzym nylonase -her, ikke å være et resultat av et rammeskift, noe som produserte et helt nytt enzym, men av to punktsmutasjoner introdusert i et eksisterende enzym, noe som forbedret dets evne til å fordøye nylonbiprodukter. Teller disse som innovasjoner? Ikke hvis du ved innovasjon mener at du gjør noe nytt, som ikke eksisterte før. Omkonfigureringen av eksisterende ting ved å bryte regulatoriske elementer eller omarrangere DNA eller endre substrat-preferanser, utgjør ikke innovasjon, i det minste ikke den type innovasjon som trengs for å forklare mangfoldet av enzymer som eksisterer. Ingen nye enzymer ble laget.
Endelig viste utviklingen av enzym nylonase -her, ikke å være et resultat av et rammeskift, noe som produserte et helt nytt enzym, men av to punktsmutasjoner introdusert i et eksisterende enzym, noe som forbedret dets evne til å fordøye nylonbiprodukter. Teller disse som innovasjoner? Ikke hvis du ved innovasjon mener at du gjør noe nytt, som ikke eksisterte før. Omkonfigureringen av eksisterende ting ved å bryte regulatoriske elementer eller omarrangere DNA eller endre substrat-preferanser, utgjør ikke innovasjon, i det minste ikke den type innovasjon som trengs for å forklare mangfoldet av enzymer som eksisterer. Ingen nye enzymer ble laget.
Vi kom ikke hit fra den ordspråkaktige, varme, lille dammen ved ikke å lage nye ting. Mange nye ting var påkrevd, inkludert 1221 nye proteinfolder (i hvertfall som nå i SCOPe -her). Det er 2008 superfamilier av proteiner, eller 4851 familier. Hvor kom de fra? Ifølge Dan Tawfik i Weizmann-instituttet: "Det vi mangler er en hypotese for de tidligere stadiene, hvor du ikke har dette spekteret av enzymatiske aktiviteter, aktive steder og folder som seleksjon kan identifisere utgangspunkt for. Evolusjonsteorien har denne (Catch-22) haken: Ingenting utvikler seg med mindre det allerede eksisterer. "Med andre ord, ifølge Tawfik, er opprinnelsen til proteiner" noe i nærheten av et mirakel."
Oversettelse og bilder ved Asbjørn E. Lund
 Hvorfor Darwinismen ikke kan fullende innovasjon eller forklare opprinnelser
Hvorfor Darwinismen ikke kan fullende innovasjon eller forklare opprinnelser Hvorfor Darwinismen ikke kan fullende innovasjon eller forklare opprinnelser
Hvorfor Darwinismen ikke kan fullende innovasjon eller forklare opprinnelser Men det er ikke i det hele tatt som ekte darwinistisk evolusjon, ved at eksperimentøren bestemmer hva det overordnede målet er (evolusjonen har ingen mål) og lager en plan for hvordan man skal oppnå det. Leder av eksperimentet bestemmer mutasjonshastigheten og forårsaker at alle disse mutasjonene finner sted i en populasjon av bakterier, samtidig, og screeningen er også designet av eksperimentet for å sortere gjennom alle de millioner av mutasjoner for ønsket , planlagt endring.
Men det er ikke i det hele tatt som ekte darwinistisk evolusjon, ved at eksperimentøren bestemmer hva det overordnede målet er (evolusjonen har ingen mål) og lager en plan for hvordan man skal oppnå det. Leder av eksperimentet bestemmer mutasjonshastigheten og forårsaker at alle disse mutasjonene finner sted i en populasjon av bakterier, samtidig, og screeningen er også designet av eksperimentet for å sortere gjennom alle de millioner av mutasjoner for ønsket , planlagt endring. skjer ved evolusjon gjennom en sti med foldede mellomliggende sekvenser." Videre, til og med et enzym med lignende struktur, men ingen delt kjemi med et annet enzym, kan ikke bli konvertert til å utføre funksjonen til det tilsynelatende relaterte enzymet.
skjer ved evolusjon gjennom en sti med foldede mellomliggende sekvenser." Videre, til og med et enzym med lignende struktur, men ingen delt kjemi med et annet enzym, kan ikke bli konvertert til å utføre funksjonen til det tilsynelatende relaterte enzymet. Endelig viste utviklingen av enzym nylonase -her, ikke å være et resultat av et rammeskift, noe som produserte et helt nytt enzym, men av to punktsmutasjoner introdusert i et eksisterende enzym, noe som forbedret dets evne til å fordøye nylonbiprodukter. Teller disse som innovasjoner? Ikke hvis du ved innovasjon mener at du gjør noe nytt, som ikke eksisterte før. Omkonfigureringen av eksisterende ting ved å bryte regulatoriske elementer eller omarrangere DNA eller endre substrat-preferanser, utgjør ikke innovasjon, i det minste ikke den type innovasjon som trengs for å forklare mangfoldet av enzymer som eksisterer. Ingen nye enzymer ble laget.
Endelig viste utviklingen av enzym nylonase -her, ikke å være et resultat av et rammeskift, noe som produserte et helt nytt enzym, men av to punktsmutasjoner introdusert i et eksisterende enzym, noe som forbedret dets evne til å fordøye nylonbiprodukter. Teller disse som innovasjoner? Ikke hvis du ved innovasjon mener at du gjør noe nytt, som ikke eksisterte før. Omkonfigureringen av eksisterende ting ved å bryte regulatoriske elementer eller omarrangere DNA eller endre substrat-preferanser, utgjør ikke innovasjon, i det minste ikke den type innovasjon som trengs for å forklare mangfoldet av enzymer som eksisterer. Ingen nye enzymer ble laget.